tg-me.com/pro_python_code/1607
Last Update:
Boltz-1 - первая доступная модель с открытым исходным кодом, которая достигает точности AlphaFold3 в прогнозировании 3D-структур белков, РНК, ДНК и небольших молекул. Boltz-1 основана на архитектуре AlphaFold3, но включает ряд модификаций, повышающих точность и общую эффективность модели.
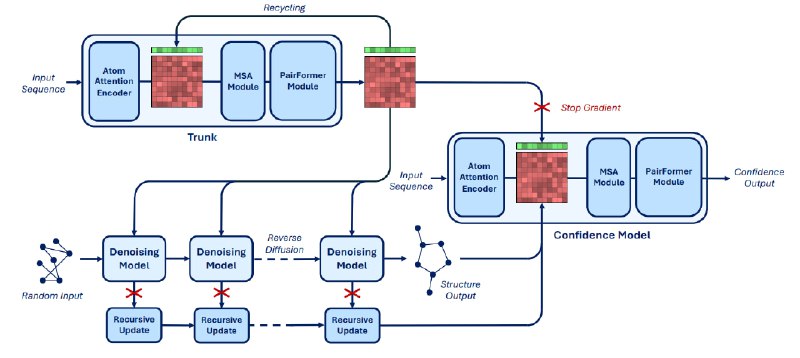
Архитектура состоит из модуля множественного выравнивания последовательностей (MSA), модуля PairFormer и диффузионной модели, работающую на двух уровнях разрешения: тяжелые атомы и токены. Токены представляют собой аминокислоты для белков, основания для РНК и ДНК, а также отдельные тяжелые атомы для других молекул.
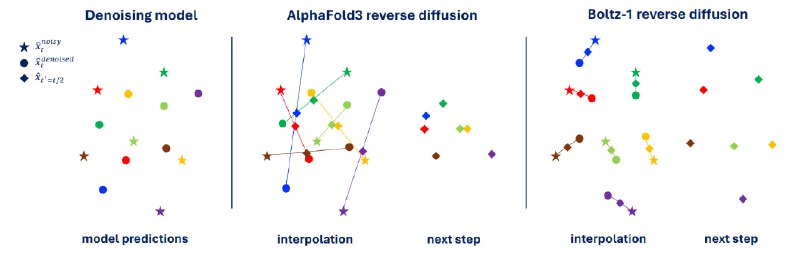
Boltz-1 использует диффузионную модель, аналогичную AlphaFold3, но Boltz-1 использует жесткое выравнивание с помощью алгоритма Кабша после каждого шага процедуры вывода, чтобы гарантировать, что интерполированная структура более похожа на очищенную от шума выборку. Это уменьшает дисперсию потерь денойзинга и предотвращает переобучение модели.
Обучение модели проводилось на структурных данных из PDB, выпущенных до 30 сентября 2021 года, с разрешением не менее 9Å. Чтобы ускорить обучение, разработчики Boltz-1 применили алгоритм сопряжения MSA с использованием таксономической информации, унифицированный алгоритм кадрирования и алгоритм определения кармана связывания. Обучение модели заняло 68 тысяч шагов с размером пакета 128, что меньше, чем у AlphaFold3.
Оценка Boltz-1 была выполнена на датасете CASP15 и на наборе PDB, специально созданном разработчиками для тестирования.
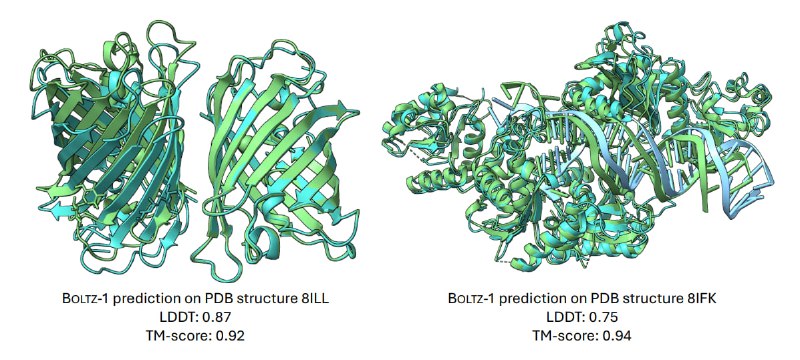
Результаты показали, что Boltz-1 сопоставима по точности с Chai-1, закрытой репликацией AlphaFold3. Обе модели демонстрируют схожие показатели среднего LDDT и среднего TM-score.
Boltz-1 продемонстрировала преимущество в предсказании взаимодействия белок-лиганд на наборе данных CASP15.
Прикладная реализация инференса, доступная в репозитории на Github, может принимать на вход форматы:
Подробные инструкции для процесса прогнозирования и дообучения опубликованы в репозитории с кодом.
# Install boltz with PyPI
pip install boltz
# run inference
boltz predict input_path
@ai_machinelearning_big_data
#AI #ML #Diffusion #3D #Biomolecular